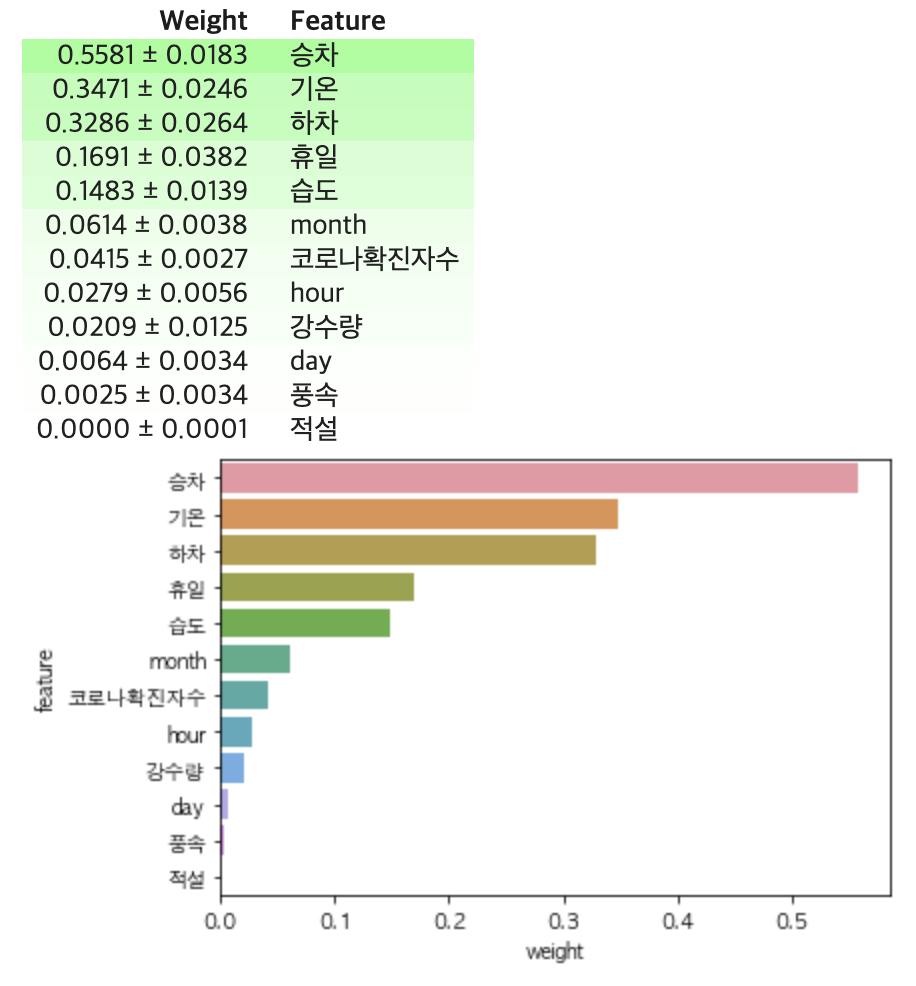
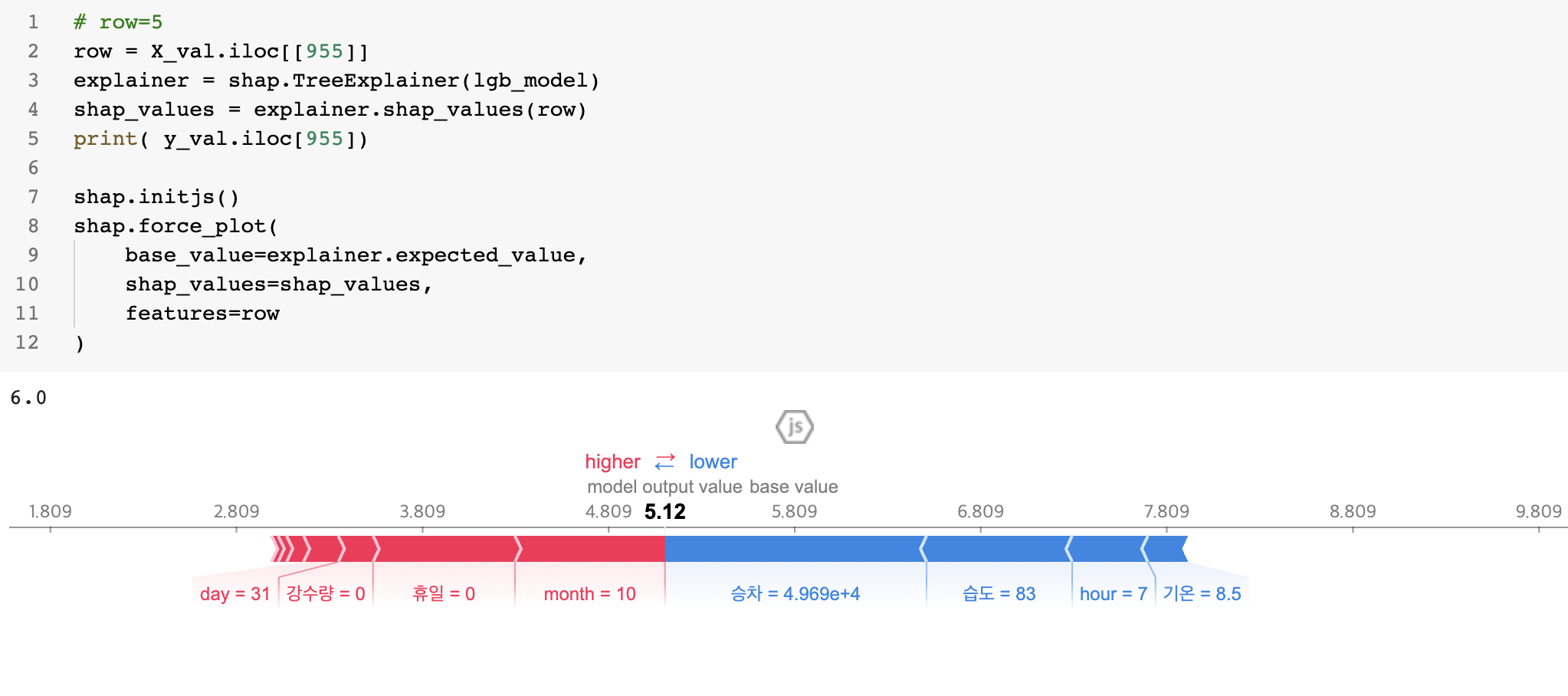
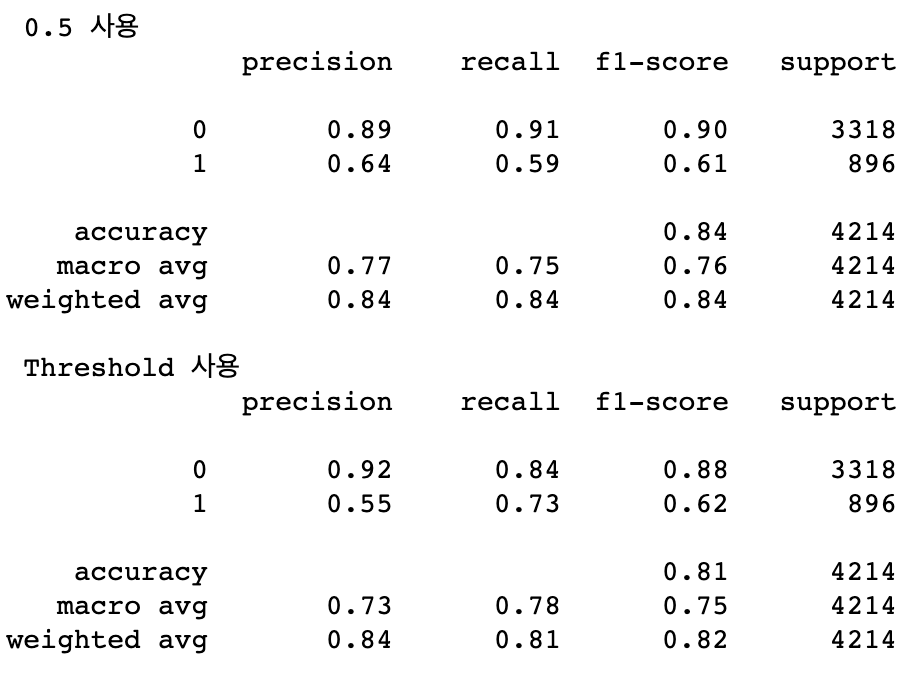
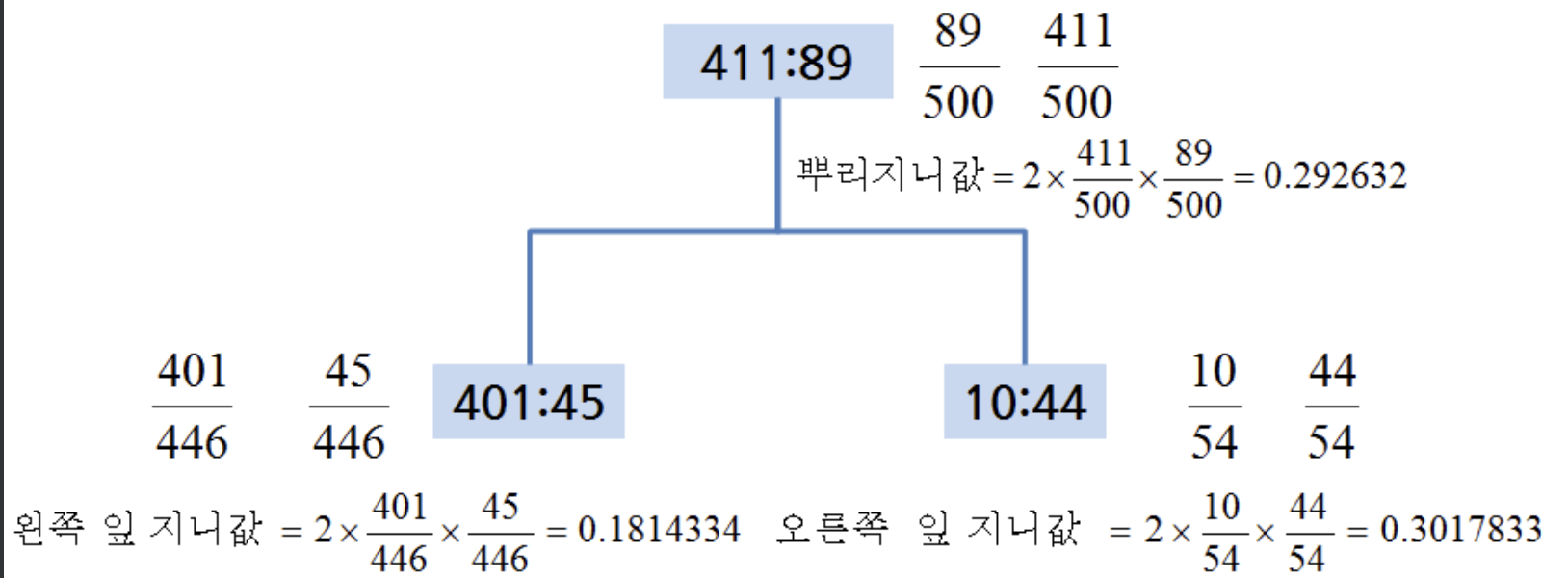
Motivation
Random Forest 가 병렬적으로 무지 많은 Decision Tree를 만든다면, 부스팅에서는 Decision Tree를 점진적으로 발전시킨 후 이를 통합하는 과정을 한다.
AdaBoost와 같이, 중요한 데이터에 대해 weight를 주는방식 vs GBDT와 같이 딮러닝의 loss function 마냥 정답지와 오답지간의 차이를 다음번 훈련에 다시 투입시켜서 gradient를 적극적으로 활용, 모델을 개선하는 방식이 있는데, XGBoost, LightGBM이 이에(후자) 속한다.
LightGBM에서 (GOSS와 EFB방식을 통해) 번들을 구성, 데이터 feature를 줄여서 학습한다
Conventional GBM need to, for every feature, scam all data instances to estimate the information gain of all the possible split points.
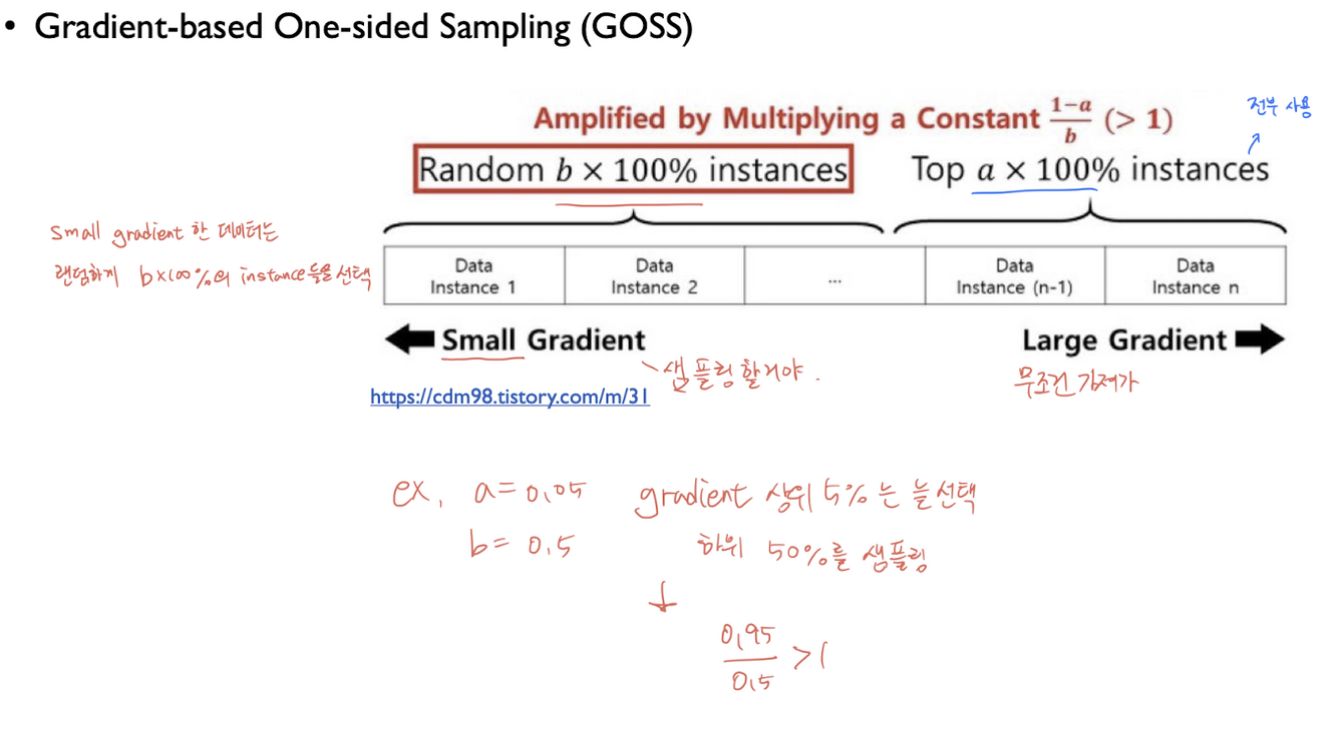
GOSS (Gradient-based One-Side Sampling)
- Data instances with different gradients play different roles in the computation of information gain
- Keep instances with large gradients and randomly drop instances with small gradients
- 각 데이터들은 서로다른 gradient를 가지며, 이에대한 information gain이 다르다 -> 어느지점을 습득하는지에 따라 다른 역할을 수행하게된다.
- large gradient한 데이터(instances)는 갖고가고, small gradient한 데이터에서 랜덤선택한다. -> 상위몇개를 고정, 하위몇개 중 랜덤하게 골라서 학습한다.
GBDT에서 모든 feature에 대해 스캔을 쭉 해서 가능한 split point를 찾아, information gain을 측정하는데, LightGBM에서는 모든 feature를 스캔하지않기위해, gradient-based One-Side Sampling 를 한다. (GOSS)
GBDT에는 weight는 없지만, gradient가 있다. 따라서 데이터의 개수를 내부적으로 줄여서 계산할 때, 큰 gradient를 가진 데이터는 그대로 사용하고, 낮은 gradient를 랜덤하게 drop하고 데이터를 가져와서, 샘플링을 해준다. gradient가 적다고 버리면 데이터 분포 자체가 왜곡될 수 있기에, 이대로 훈련하면 정확도가 낮아진다.
이를 방지하기위해 가져온 낮은 gradient 데이터에 대하여 ${1-a}\over{b}$ 만큼 뻥튀기 해준다.
(a: 큰 gradient데이터 비율, b: 작은 gradient 데이터 비율)
낮은 gradient를 가진 데이터만 drop하므로, One-Side Sampling이라고한다.
이렇게 feature를 줄여서 학습하는 것이다.

topN = a * len(I) ,전체 데이터의 a만큼 index (ex. 100개 중 a=0.2라면 topN=20)
randN = b* len(I) ,topN과 비슷하게 b만큼 index
- 모델로 일단 예측
- 실제값과의 error로 loss를 계산, weight를 계산하여 저장
- loss대로 정렬 -> sorted에 저장. sorted[1:topN] 만큼 Loss 상위 데이터들 topSet에 저장 (ex. 전체 100개 중 a=0.2라면 20개)
- 나머지 80개 중 randN개 만큼 랜덤샘플링하여 randSet에 저장 (ex. 나머지 80개 중 b=0.2라면 16개 데이터)
- UsedSet에 topSet과 randSet을 저장
- small gradient data에 fact(=${1-a}\over {b}$)만큼 weight를 부여해줌
- weak learner를 만들어서 새로운 모델로 부여 , weak leaner에는 샘플링된 데이터(UsedSet = topSet + fact assigned randSet)와 loss와 weight가 들어감
- 모델 저장
EFB (Exclusive Feature Bundling)
- In a sparse feature space, many features are (almost) exclusive, i,e., they rarely take nonzero values simultaneously (ex. one-hot encoding)
- Bundling these exclusive features does not degenerate the performance
- 하나의 객체(feature)에 대해 특정 두개의 변수가 Non-Zero값을 갖는 경우가 드물다.
- 그리하여 exclusive한 변수들을 번들링해도 성능저하가 거의 없다.

Greedy Bundling에서는 지금 현재 존재하는 feature set들에 대해서 어떠한 feature들을 하나의 번들로 묶을지 결정
Merge Exclusive Features에서는 번들링되어야하는 변수들을 이용, 하나의 변수로 값을 표현(Merge해줌)
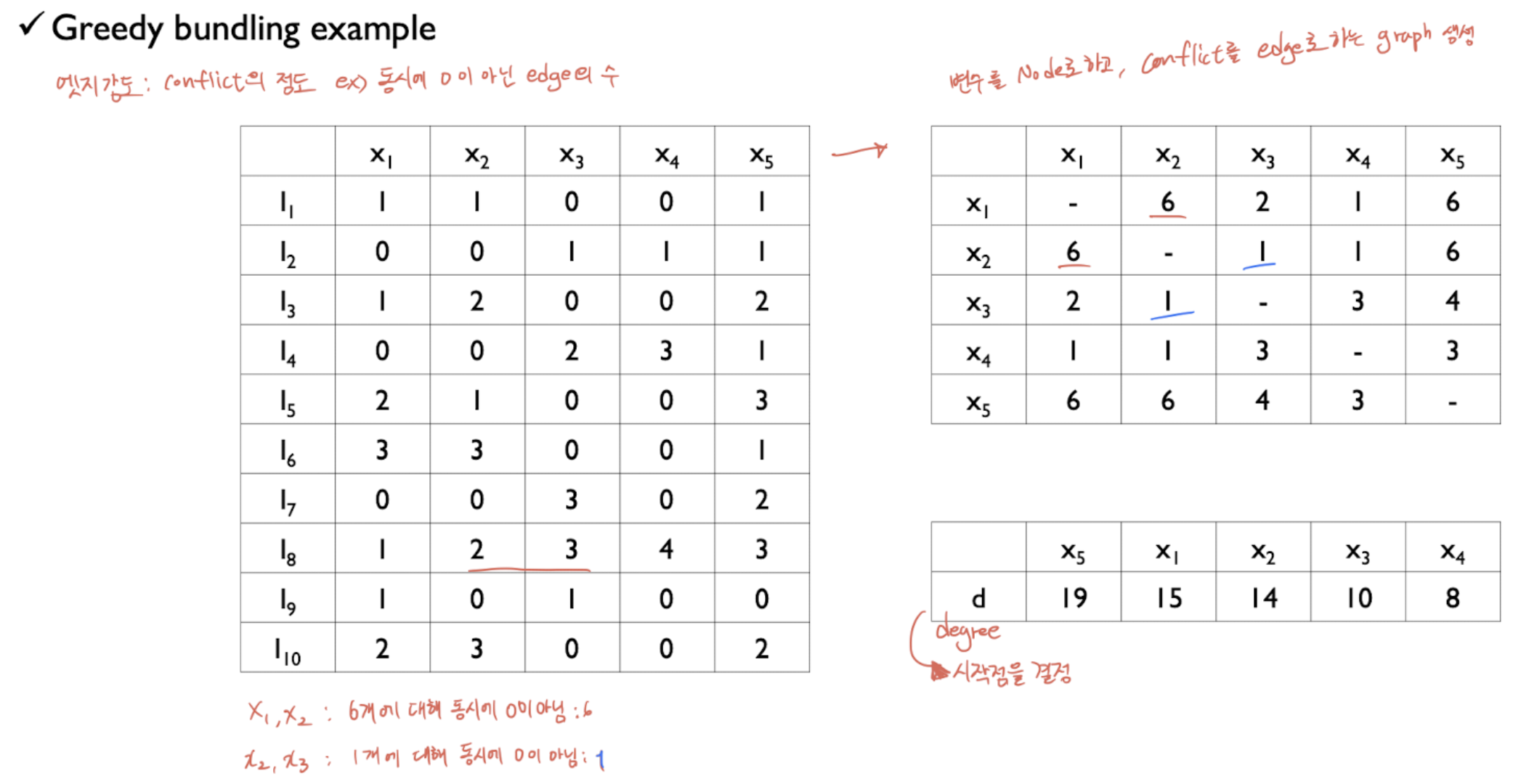
예를들어 이러한 데이터셋(x1~x5) 먼저 각각의 conflict를 Edge로 하는 Graph를 생성한다 (오른쪽 위)
여기서 conflict는 서로 상충하는 데이터 개수(동시에 0이 아닌 데이터 개수)이다.
이를 바탕으로 오른쪽 아래의 Degree를 계산할 수 있고, 이를통해 시작점을 계산한다. 위의 그림에서는 x5부터 시작.
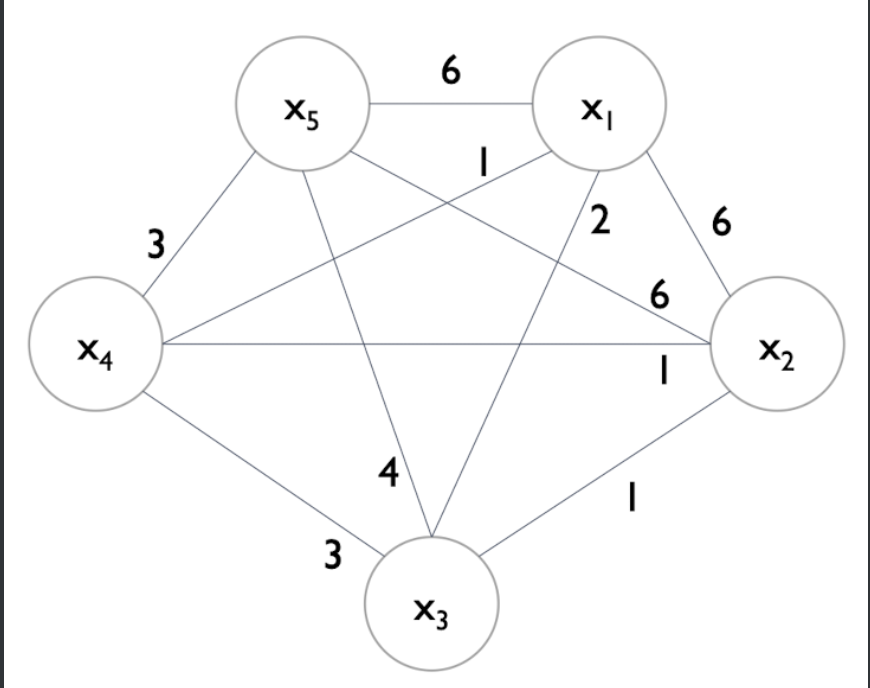
이렇게 그래프에서 x5부터 시작하며, 각각의 edge는 상충하는 데이터 개수(conflict)이다.
여기서 cut-off가 등장하는데, 이를 기준으로, 번들링에서 cut-off만큼 이상의 conflict라면, 하나의 번들로 묶지않는것이다.
위의 데이터에서는 10개 중 cut-off = 0.2로, 2개 이상 conflict 등장하면 엣지날림.
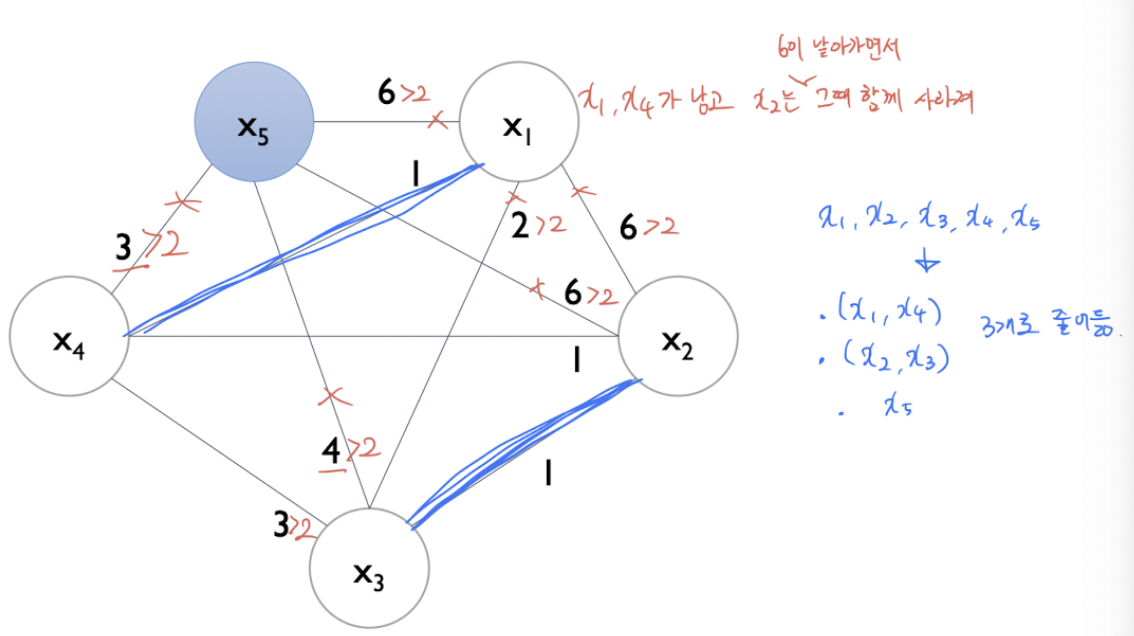
x5는 동떨어지게되므로 그대로가고,
x1을 계산할 때, x1과 x2는 6의 conflict로 날리고 x3도 날리면, ... x1,x4가 하나의 번들로 묶이게된다.
x2를 계산할 때, 이미 번들링된것을 제외하고, x3과 번들로 묶임. 더이상 묶을게 없으므로 종료.
이제 이렇게 묶은 번들에 대하여 Merge Exclusive Features를 진행한다.
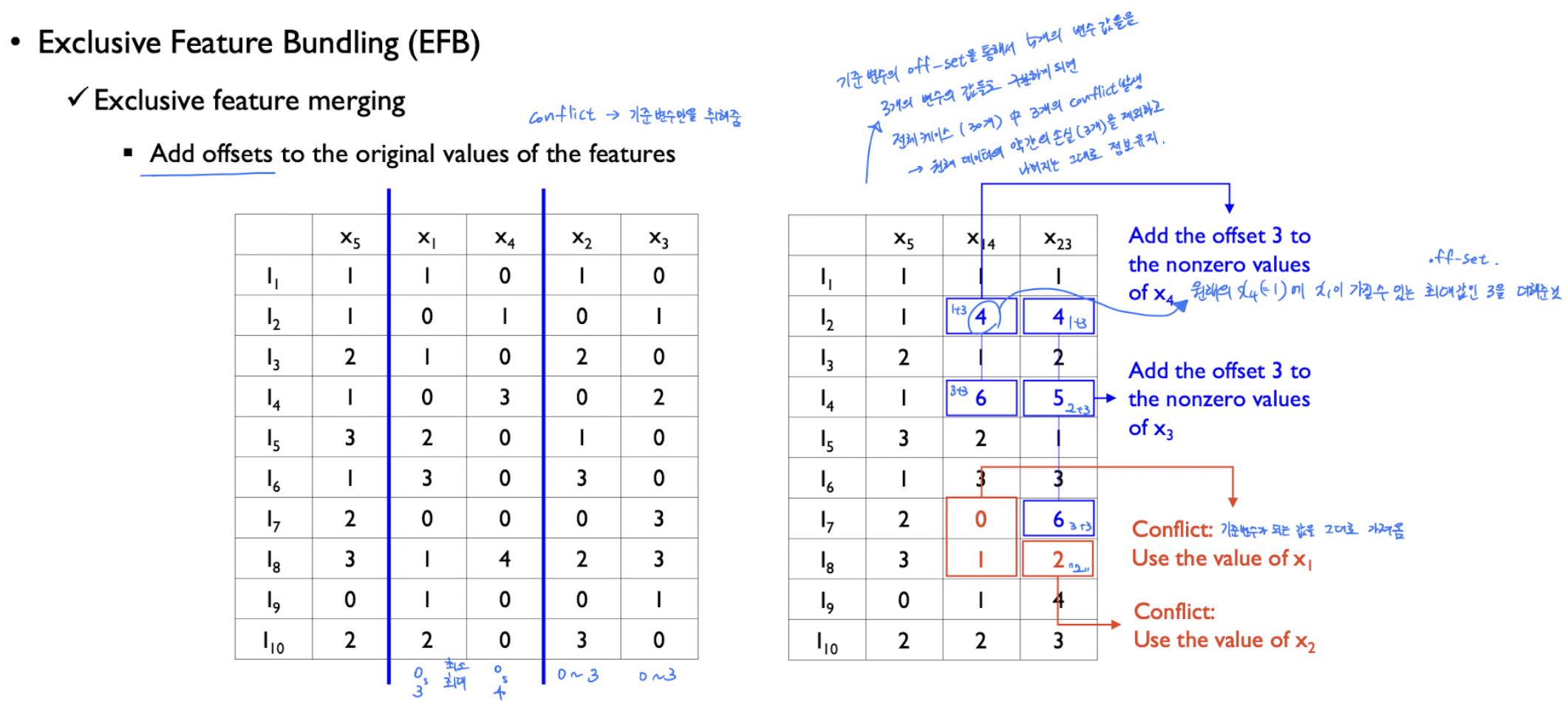
각각 column(feature)에 대해 cardinality처럼 최대와 최소값을 기록하고, 기준이 되는 feature에 함께묶인 feature와 merge를 해주는것이다.
이때, 기준값이 있다면 그대로 기준값을 사용하고, 기준값이 없다면, 기준feature의 최대값 + 묶인녀석의 값을 해서 넣게된다.
conflict한 경우(둘다 값이 있을 때 or 둘다 0일때) 기준값을 사용한다. (빨강 박스)
사용 예
사이킷런 사용
import lightgbm
lgbm = lightgbm.LGBMClassifier(n_estimators=1500, n_jobs=-1,
# scale_pos_weight=(0.888/0.111) # Unbalanced한 데이터
is_unbalance=True # Unbalanced한 데이터
)
eval_set=[(X_train,y_train),(X_val,y_val)]
lgbm.fit(X_train,y_train, eval_set=eval_set,eval_metric='f1',verbose=50 ,early_stopping_rounds=100)
lgbpred = lgbm.predict(X_val)
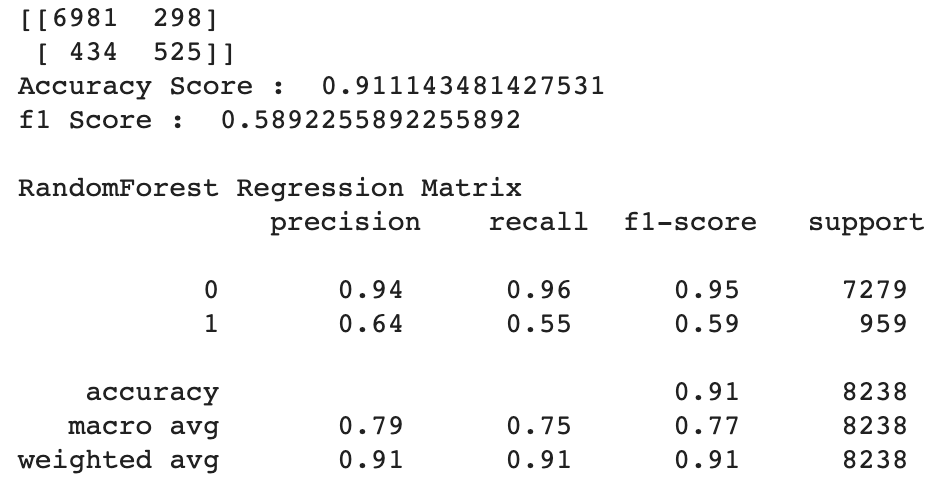
print(confusion_matrix(y_val, lgbpred))
print('Accuracy Score : ',accuracy_score(y_val, lgbpred))
print('F1 Score : ',f1_score(y_val, lgbpred))
print('\nRandomForest Regression Matrix\n' , classification_report(y_val, lgbpred))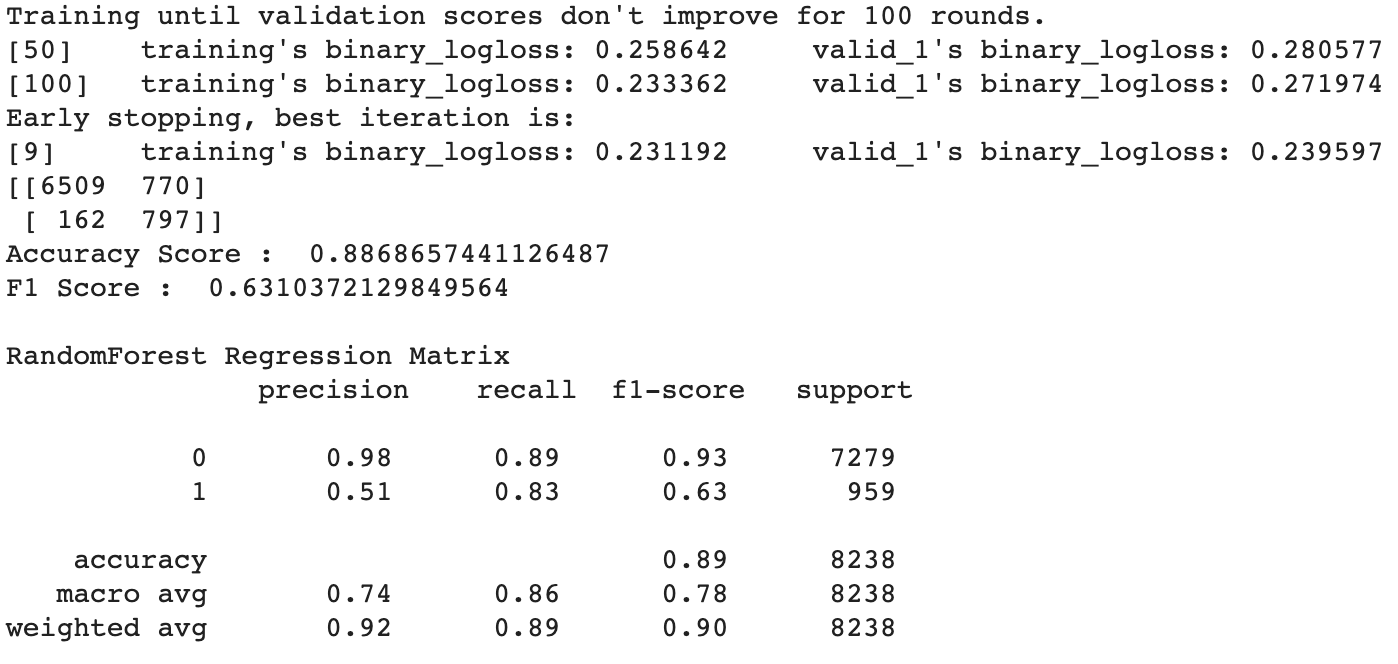
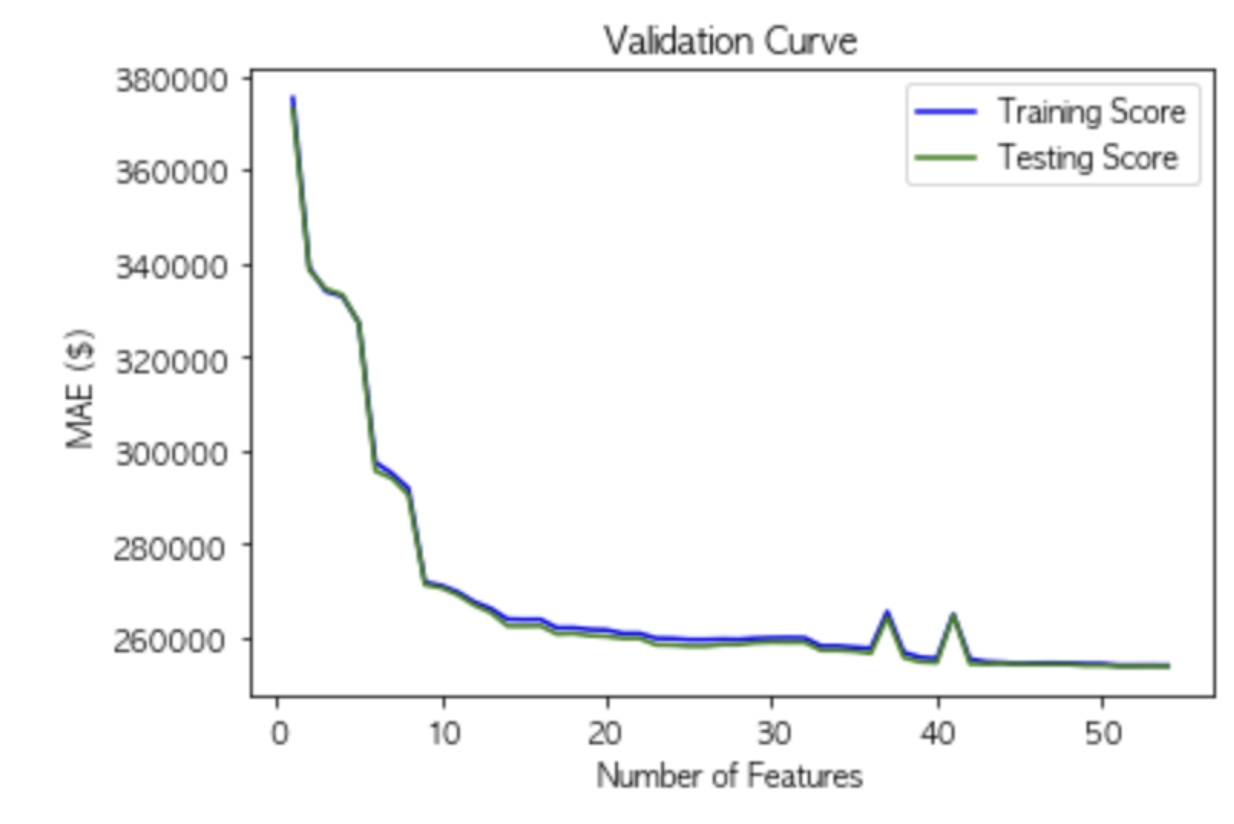
Parameter Tunning
BayesianOptimization
!pip install bayesian-optimization
from bayes_opt import BayesianOptimization
def bayesian_param_opt_lgb(X,y,init_round=15,opt_round=25,n_folds=5,random_seed=42,n_estimators=10000,learning_rate=0.05, output_process=False):
train_data=lightgbm.Dataset(data=X_train, label=y)
def lgb_eval(num_leaves, feature_fraction, bagging_fraction, max_depth, min_split_gain, min_child_weight):
params={'application':'binary','num_iterations': n_estimators, 'learning_rate':learning_rate,'early_stopping_round':100,'metric':'auc'}
params['num_leaves'] = int(round(num_leaves))
params['feature_fraction'] = max(min(feature_fraction,1),0)
params['bagging_fraction'] = max(min(bagging_fraction,1),0)
params['max_depth'] = int(round(max_depth))
params['min_split_gain'] = min_split_gain
params['min_child_weight'] = min_child_weight
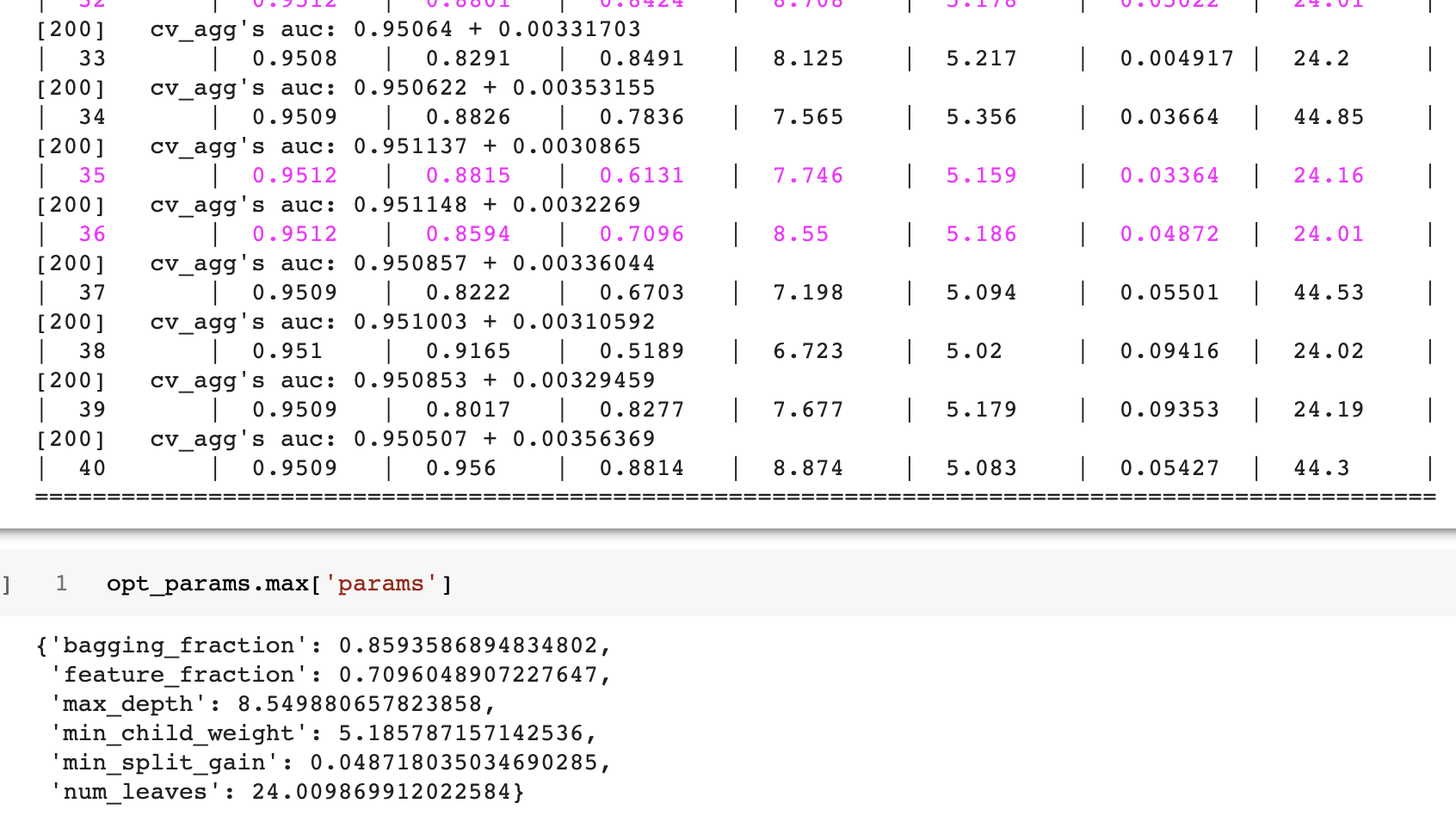
cv_result=lightgbm.cv(params, train_data, nfold=n_folds,seed=random_seed, stratified=True, verbose_eval=200, metrics=['auc'])
return max(cv_result['auc-mean'])
lgbBO=BayesianOptimization(lgb_eval, {'num_leaves':(24,45),
'feature_fraction':(0.1,0.9),
'bagging_fraction':(0.8,1),
'max_depth':(5,9),
'min_split_gain':(0.001,0.1),
'min_child_weight':(5,50)}, random_state=42)
lgbBO.maximize(init_points=init_round, n_iter=opt_round)
return lgbBO
opt_params=bayesian_param_opt_lgb(X_train,y_train)##,init_round=5, opt_round=10, n_folds=3, random_seed=42, n_estimators=100,learning_rate=0.05)
Featrue Importance를 확인해볼 수 있는 모듈을 제공한다.
from lightgbm import plot_importance
plot_importance(lgb_model)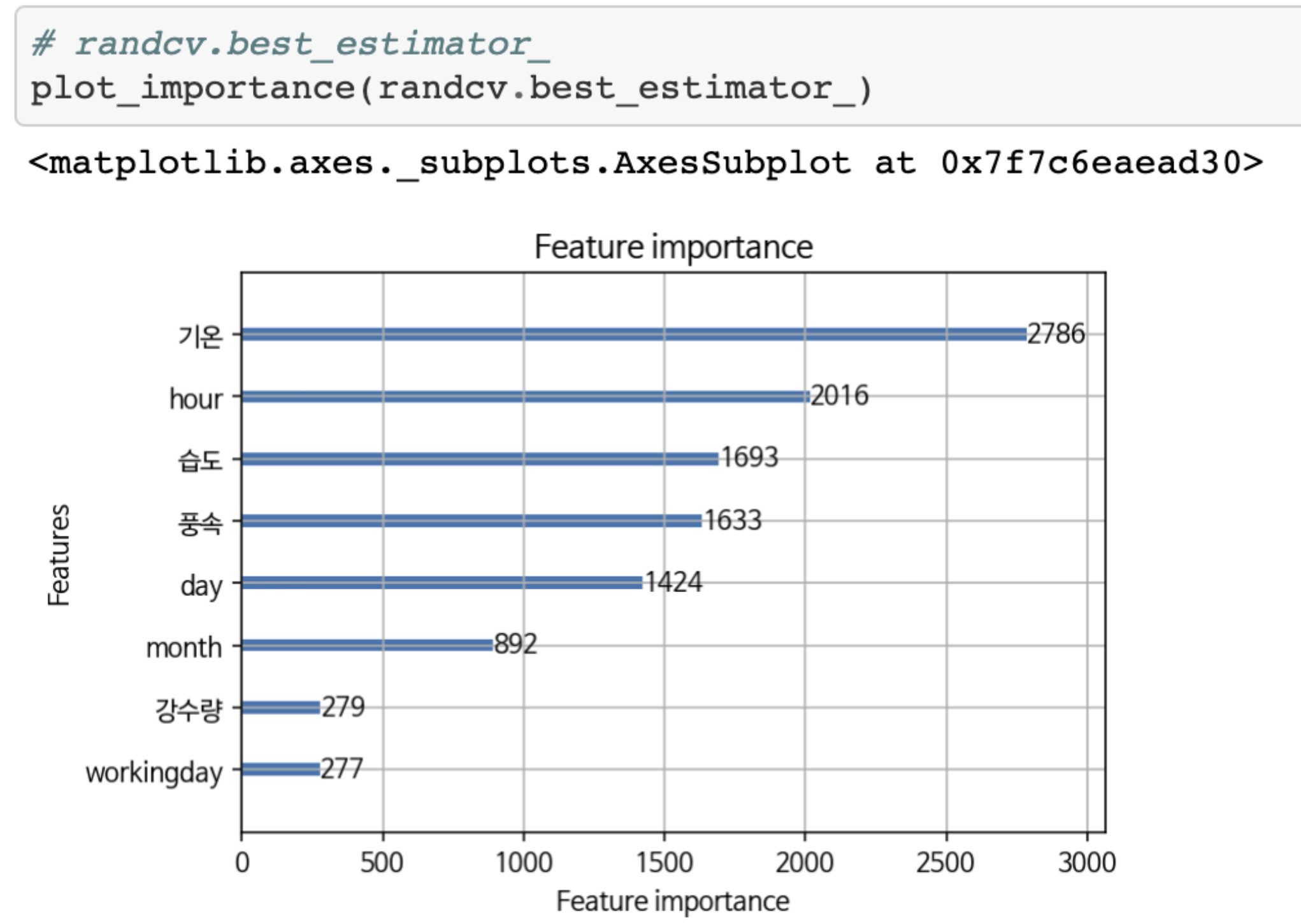
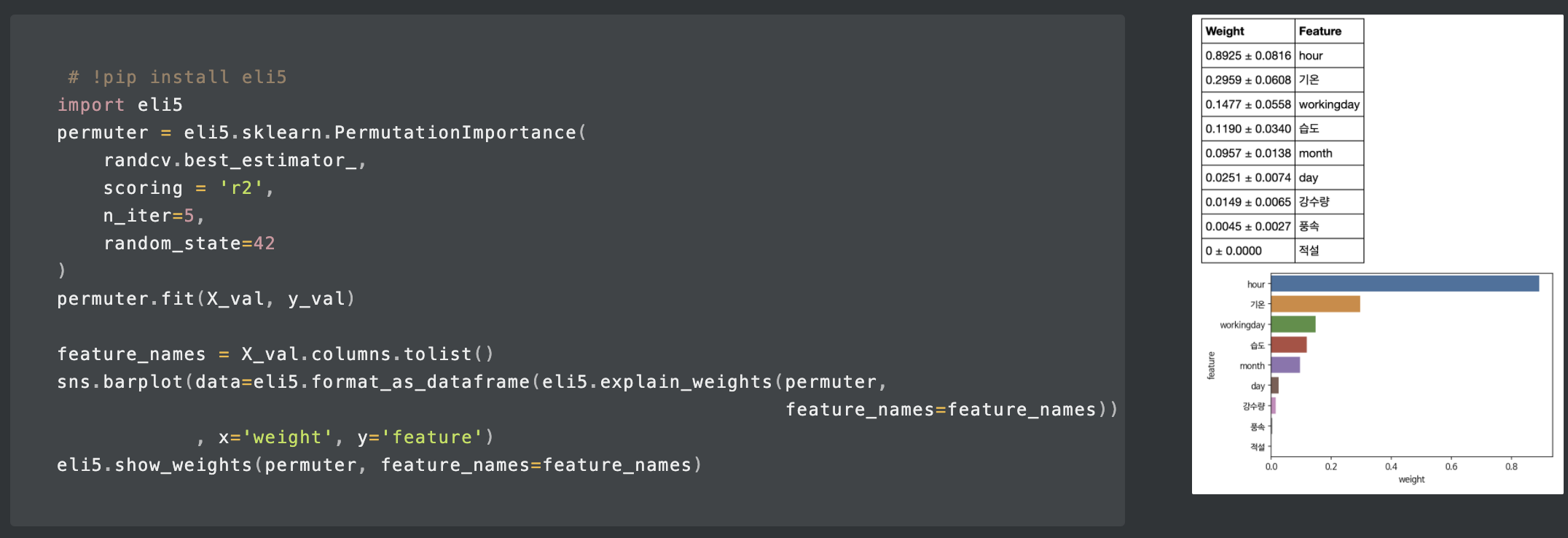
또한, eli5를 통해 Permutation Importance를 볼 수 있고, pdpbox의 pdp_plot, shap으로도 feature_importance를 볼 수 있다.
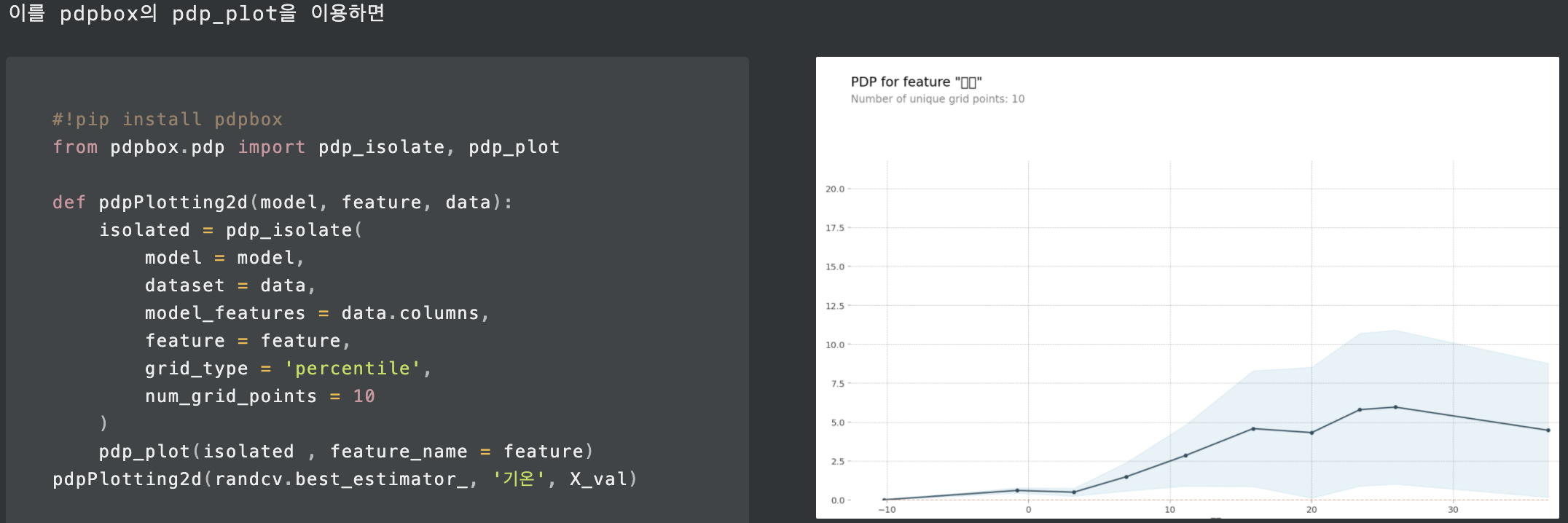
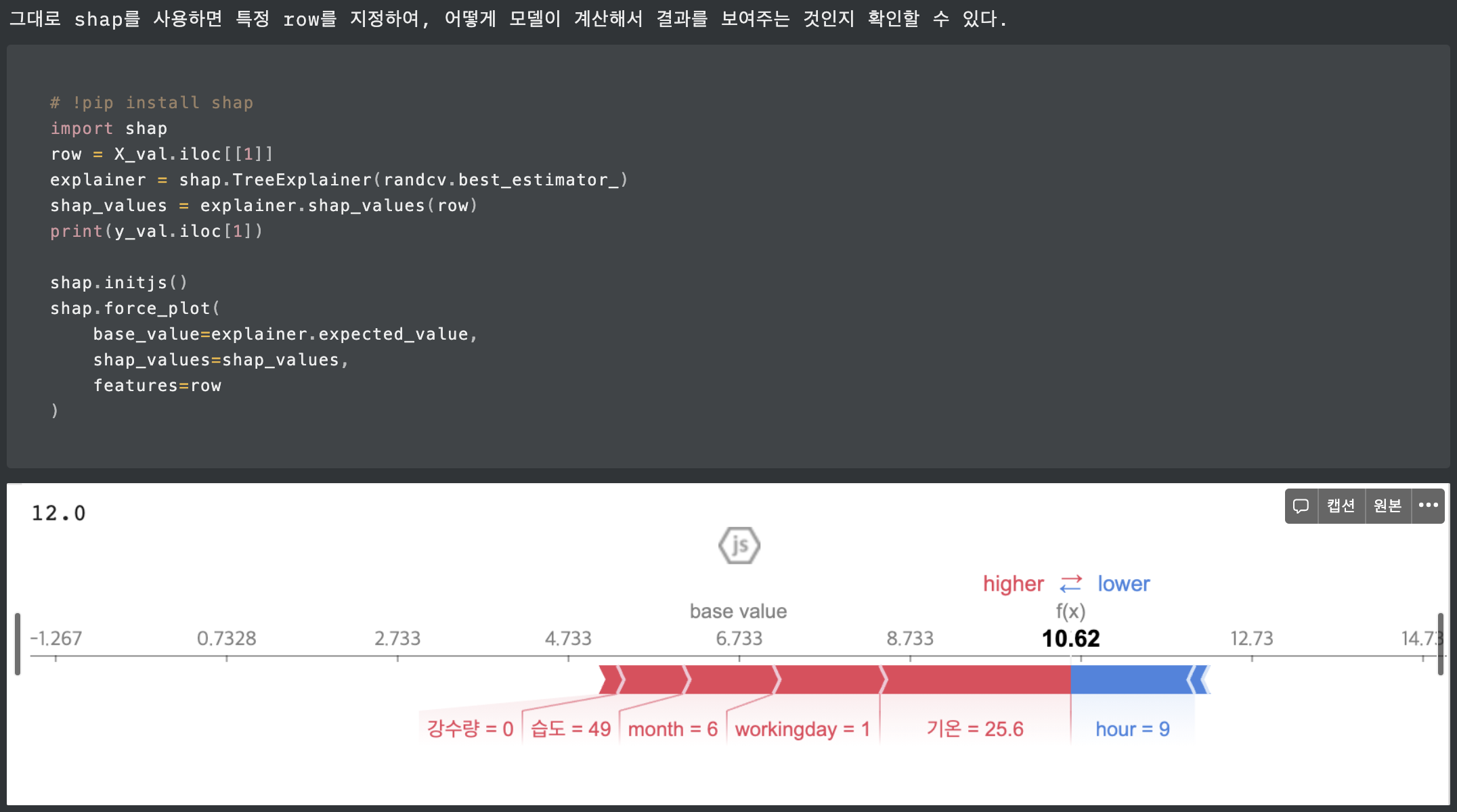
핵심 파라미터
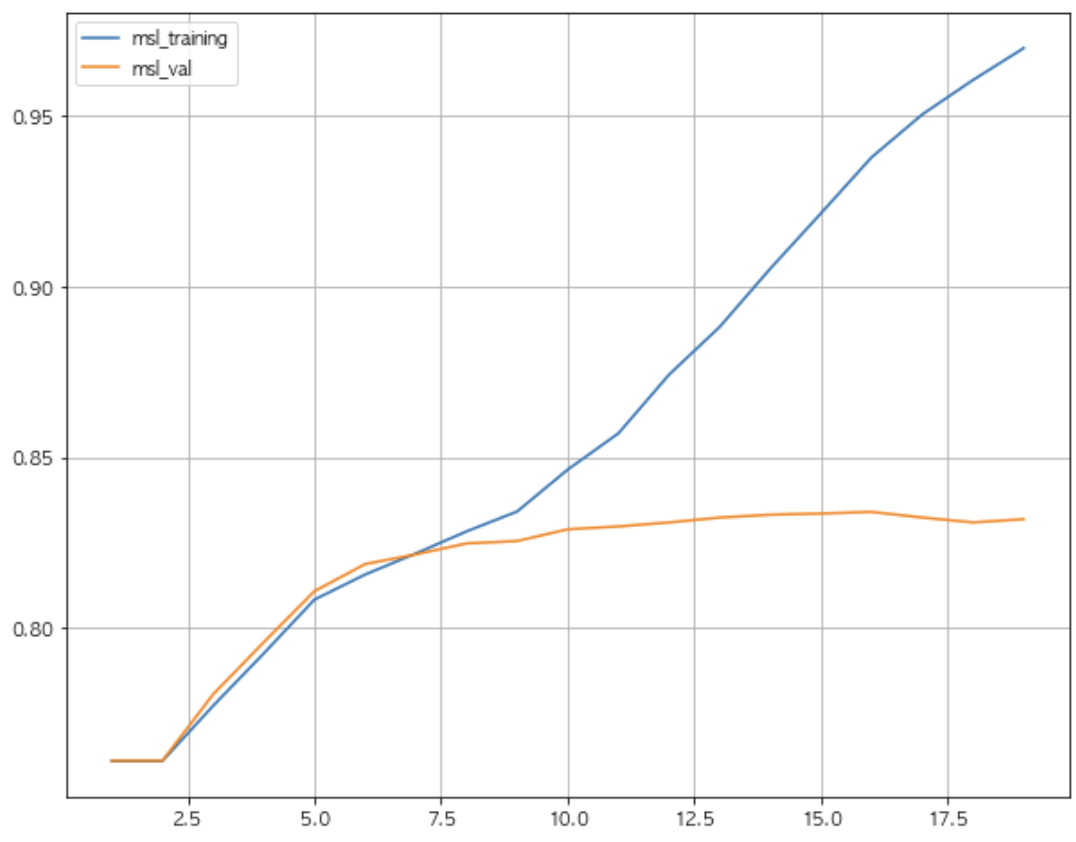
max_depth : Tree의 최대 깊이를 말합니다. 이 파라미터는 모델 과적합을 다룰 때 사용됩니다. 만약 여러분의 모델이 과적합된 것 같다고 느끼신다면 제 조언은 max_depth 값을 줄이라는 것입니다.
min_data_in_leaf : Leaf가 가지고 있는 최소한의 레코드 수입니다. 디폴트값은 20으로 최적 값입니다. 과적합을 해결할 때 사용되는 파라미터입니다.
feature_fraction : Boosting (나중에 다뤄질 것입니다) 이 랜덤 포레스트일 경우 사용합니다. 0.8 feature_fraction의 의미는 Light GBM이 Tree를 만들 때 매번 각각의 iteration 반복에서 파라미터 중에서 80%를 랜덤하게 선택하는 것을 의미합니다.
bagging_fraction : 매번 iteration을 돌 때 사용되는 데이터의 일부를 선택하는데 트레이닝 속도를 높이고 과적합을 방지할 때 주로 사용됩니다.
early_stopping_round : 이 파라미터는 분석 속도를 높이는데 도움이 됩니다. 모델은 만약 어떤 validation 데이터 중 하나의 지표가 지난 early_stopping_round 라운드에서 향상되지 않았다면 학습을 중단합니다. 이는 지나친 iteration을 줄이는데 도움이 됩니다.
lambda : lambda 값은 regularization 정규화를 합니다. 일반적인 값의 범위는 0 에서 1 사이입니다.
min_gain_to_split : 이 파라미터는 분기하기 위해 필요한 최소한의 gain을 의미합니다. Tree에서 유용한 분기의 수를 컨트롤하는데 사용됩니다.
max_cat_group : 카테고리 수가 클 때, 과적합을 방지하는 분기 포인트를 찾습니다. 그래서 Light GBM 알고리즘이 카테고리 그룹을 max_cat_group 그룹으로 합치고 그룹 경계선에서 분기 포인트를 찾습니다. 디폴트 값은 64 입니다.
출처 :
LightGBM
Go Lab
고려대 일반대학원 산업경영공학과 비즈니스 애널리틱스 강의 (Pilsung Kang교수님)
파라미터튜닝
https://lsjsj92.tistory.com/548?category=853217
https://lightgbm.readthedocs.io/en/latest/Python-Intro.html#setting-parameters
http://machinelearningkorea.com/2019/09/25/lightgbm의-핵심이해/
'Machine Learning > Boosting' 카테고리의 다른 글
| XGBoost_2_Output Value (0) | 2021.03.03 |
|---|---|
| XGBoost_1_트리만들기, Similarity Score, Gain, Pruning(Gamma) (0) | 2021.03.03 |